1. 引言
MicroRNAs (miRNAs)广泛存在于哺乳动物细胞中。它们是长度约为22 nt的内源性非编码RNA。miRNA参与转录后基因表达的调控,从而调控细胞生长和组织分化,与生命过程中的发育和疾病有关 [1] [2] [3]。研究表明,最初的miRNA是在20年前被发现。从那时起,成千上万的miRNAs从一系列物种中被发现 [4] [5]。此外,越来越多的研究表明,miRNA在生物生命发育过程的多个阶段中发挥着关键作用 [6],例如细胞生长、增殖 [7]、发育 [8],分化 [9],凋亡 [10],老化 [3] 等。研究人员使用生物实验方法来建立疾病和miRNA之间的关联,这些方法成本高、周期长且容易失败。因此,我们需要开发一个全新的计算模型框架来识别miRNA与疾病之间的关联。
有许多算法通过构建复杂的异构网络来预测miRNA与疾病的关联,Jiang等人集成多种算法和生物数据,以及神经网络机器学习 [11]。构建miRNA功能相似性网络矩阵和已知人类疾病-miRNA网络矩阵,然后计算网络矩阵中节点的相似度得分,得分越高,与疾病相关的可能性就越大。Shi等人 [12] 考虑了多种因素,将疾病基因与miRNA靶基因之间的功能联系添加到蛋白质相互作用(PPI)网络中,建立miRNA-疾病关联网络,采用随机游走法构建miRNA-疾病关联预测方法。Mørk等人 [13] 提出了一种miRPD方法来获得疾病蛋白质与miRNA通过组合三个关联,miRNA与蛋白质之间的关联评分矩阵,蛋白质与疾病之间的关联评分矩阵,miRNA与疾病之间的共享蛋白质评分矩阵。Chen等人 [14] 提出第一个基于全局网络相似度的计算模型RWRMDA,是基于经过验证的miRNA与疾病之间的关联信息和人类miRNA功能相似性信息,采用随机游走方法,RWRMDA通过对几种关键癌症的交叉验证实现了出色的预测性能。然而,它有局限性,即它不能用于miRNA与疾病之间的未知关联。Chen等人 [15] 还提出了一种名为WBSMDA的方法,通过整合miRNA的功能相似性、疾病的语义相似性、miRNA的高斯核相似性和疾病以及miRNA与疾病之间的已知相关性来计算最终相关性评分。WBSMDA可以有效地识别未知疾病-miRNA关联。Chen等人 [16] 又开发了一种新的算法模型HGIMDA,其性能优于上述四种计算算法(WBSMDA、RLSMDA、RWRMDA和HDMP)。
深度学习、机器学习和神经网络也广泛应用于生物信息学的预测和判别实验。Xu等人 [17] 基于miRNA-靶标相互作用提取特征,提出miRNA-靶标失调网络(MTDN)并使用SVM分类器区分阳性或阴性样本。Chen和Yan [18] 提供这RLSMDA揭示疾病与miRNA之间的关系,RLSMDA可用于没有已知相关miRNA的疾病。此外,它是一种半监督(不需要负样本)和全局的方法(同时优先考虑所有疾病关联)。Chen [19] 为了进一步提高miRNA-疾病关联(RBMMMDA)的预测性能,开发了受限玻尔兹曼机器模型,它可以有效地预测不同类型的miRNA和疾病。
本研究试图将嵌入式学习功能与从未知关联中查找关联相结合。我们的方法由三部分组成:疾病嵌入模型、miRNAs嵌入模型和深度稀疏自动编码模型。我们使用深度学习算法来构建这些模型。首先,我们尝试训练我们的疾病嵌入模型,同时训练miRNA嵌入模型来学习它们在高维空间中的表示。然后我们通过疾病和miRNA的嵌入模型将已知的已验证的miRNA-疾病关联起来,然后通过深度稀疏自动编码器模型来学习潜在的特征。我们研究的主要贡献如下:
1) 我们向自动编码器添加稀疏性,该方法向重构误差添加稀疏性惩罚,以限制并非隐藏层中的所有单元在任何时候都被激活。
2) 我们实施了一种嵌入式学习方法来提取疾病和miRNA的表示。通过将疾病语义相似度与疾病高斯相互作用谱核相似度、miRNA功能相似度和miRNA高斯相互作用谱核相似度相结合,自动学习高维密集向量特征来表示疾病和miRNA。
2. 数据准备与计算框架
2.1. 人类miRNA与疾病的关联
我们采用人工收集和编译的miRNA-疾病关联数据的数据库作为基准数据集。直接下载已知的人类miRNA和疾病的相关数据(http://www.cuilab.cn/static/hmdd3/data/hmdd2.zip)。
2.2. 疾病语义相似性
从国家医学网络图书馆(http://www.nlm.nih.gov)下载包含人类疾病的C类MeSH。构建有向无环图(DAG)旨在计算疾病的语义相似度 [20]。对于某些疾病节点D,定义计算公式为:
,其中
包括疾病节点D及其前辈的节点集,
表示子节点与父节点直接链接的边集。疾病D的语义值定义如下:
(1)
(2)
其中,
表示DAG(D)中每个节点D对疾病D的语义价值贡献。在DAG(D)中,疾病D是对自身最具体的描述,其语义贡献应该最大,设置为1;距离疾病D较远的节点是疾病D的更一般描述,因此对疾病D的语义价值贡献较小。Δ为对语义贡献值的衰减因子(0 < Δ < 1,在本研究中,Δ设置为0.5)。假设当任何两种疾病的DAG中有更多重叠时具有更高的语义相似度,计算疾病
与
之间的语义相似度得分定义如下:
(3)
DAG(D)中节点D对疾病D语义值的定义如下:
(4)
同理,定义疾病D的语义值DV2(D)以及疾病与疾病的相似度如下:
(5)
(6)
结合两种疾病的语义相似度计算结果,疾病的语义相似度如下:
(7)
2.3. miRNA功能相似性
基于具有相似功能的mirna更倾向于与相似的疾病表型相关的假设,我们使用Wang等人提出的方法 [20] 计算miRNA的功能相似性。
直接从http://www.cuilab.cn/files/images/cuilab/misim.zip下载。功能相似性矩阵(FS)包含383 × 383个miRNA,
表示miRNA
与
之间的相似度值。
2.4. miRNA与疾病高斯相互作用谱核相似性
基于已被验证的miRNA-疾病关联矩阵,引用了miRNA的高斯相互作用谱核相似度和疾病的高斯相互作用谱的核相似度。
为miRNA
与
之间的高斯相互作用谱核相似度。类似地,
表示疾病
与
之间高斯相互作用谱核相似性。
2.5. 计算框架DSAEMDA
DSAEMDA由三个主要部分组成:疾病模型、miRNA模型和深度稀疏自动编码器模型,其中自动编码器包括三层编码器(用于在高维空间中编码已知的miRNA-疾病关联)和三层解码器(用于重建计算误差)。经过验证的miRNA-疾病已知关联用于训练深度稀疏自动编码器。DSAEMDA工作流程如图1所示。
3. 特征表示
在基于神经网络的计算方法中,疾病和miRNA的正确表示非常重要,对模型的预测有很大影响。Peng等人引入基因层来计算miRNA基因网络和疾病基因网络中的关联分数,以生成疾病(或miRNA)和基因的Pearson相关性为载体,表达疾病(或miRNA) [21]。Xuan等人结合miRNA和疾病相似性及其关联形成特征表征 [22],miRNA矩阵的表示和疾病矩阵的表示,通过整合miRNA功能相似性矩阵、已知的miRNA-靶基因相互作用矩阵以及经过验证的miRNA-疾病关联矩阵来提取miRNA特征和疾病特征 [23]。
与上述方法不同,我们应用学习算法通过整合两种疾病相似性和疾病的高斯相互作用谱核相似性来直接提取疾病特征表示。miRNA的表示来源于整合miRNA的功能相似性和miRNA的高斯相互作用谱核相似性。我们将疾病映射到高维疾病向量空间,同时将miRNA映射到高维miRNA向量空间,构建miRNA回归模型和疾病回归模型,通过在高维空间中的距离来学习和表示这些准确且信息丰富的向量。
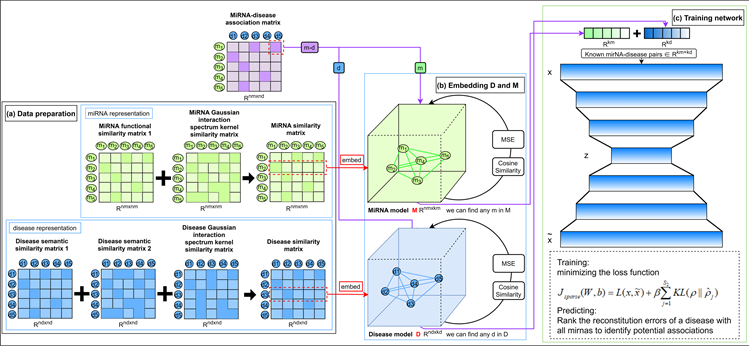
Figure 1. DSAEMDA model framework for predicting potential miRNA-disease associations. It includes three parts: (a) data preparation contains disease data and miRNAs data; (b) data embeddings to learn miRNA and disease representation features in high-dimensional spaces; (c) a deep sparse autoencoder for predicting the associations between miRNAs and diseases
图1. DSAEMDA模型框架用于预测潜在的miRNA-疾病关联。包含三个部分:(a) 数据准备阶段包含疾病数据和miRNA数据;(b) 数据嵌入学习miRNA和疾病表示在高维空间中的特征;以及(c) 用以预测潜在miRNA-疾病潜在关联的深度稀疏自动编码器
3.1. 疾病特征表示
神经语言处理(NPL)可以自动学习用向量来表示不同维度的单词或句子 [24] [25] [26],我们训练两个高维模型来学习疾病的和miRNA的表示。
我们首先为每种疾病定义唯一的数字来表示特定的疾病,例如,
表示矩阵D的第i行的密集向量是从矩阵D的嵌入中检索出来的。在训练之前,D中的元素是不确定的,我们随机初始化D中的元素。经过多次学习迭代、最小化误差和反向传播,D被训练为高维空间中的有效疾病表达。D的定义如下:
(8)
其中
代表第i个疾病的高维空间向量。nd是疾病个数,kd是疾病映射在高维空间中的向量大小,所以疾病矩阵在高维空间的表示为
。
单独使用疾病语义相似度矩阵很难获得很好的预测性能。同时,仅使用疾病高斯相互作用谱核相似性来预测已知的miRNA-疾病关联计算也是不够准确的。因此,为了获得良好的预测性能,需要将疾病高斯相互作用谱核相似度矩阵KD与疾病语义相似度矩阵SS结合起来。通过对两个矩阵进行加权整合,我们将其表示为SD。最后,我们利用矩阵SD来学习高维空间中疾病的向量D。疾病矩阵如下:
(9)
SD、
、KD维度相同;换句话说,是介于0和1之间的权重。疾病的高斯相互作用谱核相似度矩阵KD和疾病语义相似度矩阵SD中的每个元素都在(0, 1)的范围内。我们将
视为任意两种疾病
和
之间的高维空间距离。根据余弦相似度的计算特性,通过两个向量之间的夹角余弦来衡量两个向量之间的相似度 [27] [28]。对余弦函数的公式进行算术调整,保证任意两种疾病的计算结果在(0, 1)内。疾病模型的余弦相似度SD'由以下公式给出:
(10)
我们的疾病模型通过构建回归模型来学习高维空间中疾病的特征。任意两种疾病在高维空间向量的分值越高,意味着它们在高维空间中具有更高的相似性。疾病计算模型最小化所有疾病之间的损失函数定义如下:
(11)
是我们训练样本的总数。在每次训练迭代中,以均方损失为准则,通过反向传播的独立自适应学习率(Adam)算法更新疾病矩阵D。
3.2. miRNA特征表示
类似地,代表495个miRNA的高维空间矩阵M定义如下:
(12)
其中
代表第i个miRNA的高维空间向量。nm是miRNA个数,km是miRNA映射在高维空间中的向量大小,所以miRNA矩阵在高维空间的表示为
。同样,我们通过以下等式学习矩阵M:
(13)
其中
是训练样本总数,SM和SM'的定义公式如下:
(14)
(15)
4. 基于自动编码器的关联预测器
4.1. 自动编码器
Autoencoder简称“AE”,AE是Hinton [29] 在1980年代提出的一种无监督聚类算法。它已广泛应用于特征提取、数据压缩、特征降维、异常检测和模型生成。神经网络中的自动编码器是一种无监督学习算法,也就是说,它不需要分类标签。它使用反向传播最小化损失函数算法使目标输出值无限接近原始输入值。基本的自动编码器是三层神经网络模型,第一层是数据输入层,第二层是隐藏层,第三层是输出重建层。它也是一种无监督学习模型。我们的七层稀疏自动编码器模型主要由两个元素组成:编码器(用于数据压缩和特征提取)和解码器(用于重构输入)。编码器的任务是压缩数据并从高维模型D和M中提取特征,解码器的任务是恢复数据并从隐藏码中重构输入。我们的神经网络模型由七个完全连接的层组成。图2显示了我们的7层深度稀疏自动编码器DSAEMDA的架构。
miRNA与疾病之间的相关性由重建误差表示。重构误差的程度直接反映了miRNA与疾病的相关性。我们的稀疏自编码器输入是疾病dis和miRNA mir之间的链接向量,链接向量主要由两部分组成,一部分是查询疾病模型D中的疾病高维向量dis,另一部分是查询miRNA模型M中的miRNA高维向量mir,接下来,向量dis和向量mir链接(dis, mir)作为输入向量到我们的深度稀疏自动编码器。
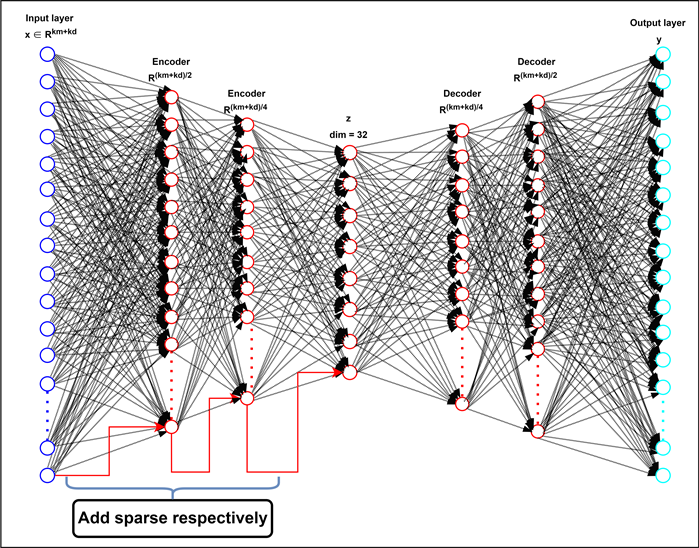
Figure 2. The framework of sparse autoencoder
图2. 稀疏自动编码器框架
第i个训练样本定义如下:
(16)
样本
,编码器通过以下公式压缩数据并提取潜在代码的特征
:
(17)
(18)
,L = 3表示编码器的隐藏层数为3。
表示l隐藏层,
表示输入
。权重矩阵
和偏差
是l层的参数。
是编码器层的输出,代表
的潜在表示。通过非线性激活函数
(ReLU)校正线性元素 [30]。
解码器旨在从编码器的潜在表示
中尽可能多的重建输入
。下面的公式定义了解码器:
(19)
(20)
其中
表示l层隐藏层,解码层的第一层为中间隐藏层
。,L = 3表示解码器的隐藏层数为3,权重矩阵
和偏差
是解码器l层的参数。我们模型中最后一个解码器的输出
是对输入
的重构。将
和
分别设置为ReLU和TANH。
4.2. 稀疏性
受机器学习领域的启发 [31]。为了给自编码器添加稀疏性,我们选择的方法是添加KL散度,KL散度就是相对熵 = 交叉熵 − 信息熵,在重构误差中添加稀疏性惩罚,以限制隐藏层中的所有单元在任何时候都被激活。
我们用
来表示在给定输入x下自编码神经网络中隐藏神经元j的激活程度。隐藏神经元j的平均激活度(在训练集上平均)表示为:
(21)
稀疏性限制可以理解为最小化隐藏层神经元的平均激活度,可以表示为
,其中
是稀疏性参数,通常是接近于0的较小值,我们在神经网络优化的原始目标函数中加入稀疏性限制作为额外的惩罚因子,我们可以选择如下形式的惩罚因子:
(22)
其中
为在隐藏层中神经元的数量,索引j依次代表隐藏层中的每一个神经元。也可以将其描述为相对熵,表示为:
(23)
是两个伯努利随机变量之间的相对熵,均值为
和
。
现在,我们的总成本函数可以表示为:
(24)
定义如下:
(25)
N是已知已验证的miRNA-疾病关联的总数。前面的第一项是平方损失,后面的第二项是正则化的Frobenius范数 [32],
和
是两个超参数。训练我们的深度稀疏自编码器的目标是尝试最小化上述损失函数并迭代更新神经元参数。
5. 数据实验与结果分析
5.1. 实施细节
在我们的环境中使用的是一个开源机器学习框架PyTorch,版本为1.7.1。我们的实验是在配备NVIDIA1080TI图形处理器的Windows10平台上进行的。
DSAEMDA根据以下程序进行训练。我们首先使用均方误差(MSE)作为损失函数来训练疾病模型和miRNA模型。在100个epoch之后,我们获得疾病D和miRNA M的两个高维空间矩阵模型,然后将miRNA与疾病的已知关联嵌入到D和M中来训练深度稀疏自编码器。我们的深度稀疏自编码器通常会在100~150 epoch收敛。
HMDDv2.0数据库用于训练深度稀疏自编码器,设置疾病数量nd为383,miRNA数量nm为495。权重初始化时,当初始化时,两个模型的权重都均匀地分布在−0.1和0.1之间。然后利用PyTorch中实现两个嵌入学习模型。最后生成疾病矩阵模型D和miRNA矩阵模型M,分别表示疾病和miRNA的高维模型。疾病模型通过带有反向传播的Adam方法进行端到端训练。Adam算法用于优化上述模型 [33]。
,
,
,batch = 128。初始学习率 = 1e−4,如果损失没有改善,每4步学习率降低到原来的十分之一。最后,最小化损失函数迭代优化得到最佳模型。在实验参数设置中,设置了kd = km。相同的参数设置被用于训练miRNA高维空间模型。
对于深度稀疏自编码器模型,模型的输入是已知已验证的miRNA与疾病关联的串联高维空间向量,表明输入层初始神经元个数为(kd + km)。第一层编码器的输出为(kd + km)/2作为第二层编码器的输入,第二层编码器的输出为(kd + km)/8作为第三层的输入编码器,中间隐藏码的大小为32。解码器的结构与编码器的结构相反。
5.2. 评价结果
在我们的能力范围内,为了得到一个可靠和稳定的模型,我们使用五折交叉验证(5-CV)来评估我们模型的推理能力。已知的miRNA-疾病关联被用来训练我们的模型,而未知关联不参与我们的训练过程。
在5-CV中,将已知的miRNA-疾病关联被随机分成5组,一组作为测试样本,其余四组作为训练样本。该步骤重复五次作为一个完整的循环。为了获得公平公正的数据,我们为每一轮训练样本训练了一个深度稀疏自动编码器。接下来,分别计算未知miRNA-疾病关联的重建误差和测试样本的重建误差。最后,我们根据所有重建误差进行排名。重复这些步骤5次的目的是为了减少随机样本分割带来的偏差,并将平均排名作为我们模型实验的最终结果。
此外,AUC定义为ROC曲线下的面积。AUC值用作我们模型的评估标准。因此,DSAEMDA获得的5-CV方案的AUC为0.9412 (表1)。为了进一步证明DSAEMDA模型预测的优越性,我们还将DSAEMDA与AEMDA [34]、GRL21NMF [35]、ICFMDA [36]、SACMDA [37] 和IMCMDA [38] 进行比较。在5-CV方案下,AEMDA、GRL21NMF、ICFMDA、SACMDA,和IMCMDA模型的AUC,分别为0.9383、0.9276、0.9045、0.8763和0.8330,表明DSAEMDA的性能优于其他模型。可以说SAEMDA模型在预测miRNA-疾病潜在关联方面取得了显著进展。

Table 1. Comparison between DSAEMDA and other prediction methods
表1. DSAEMDA与其它预测方法比较
6. 结论
在我们的研究中,我们为自动编码器添加了稀疏性,并为预测miRNA-疾病潜在关联创建了一个新的预测框架,称为DSAEMDA。在疾病方面,将两种不同疾病的语义相似度矩阵与疾病的高斯相互作用谱核相似度矩阵相结合。在miRNA方面,将miRNA的功能相似矩阵与miRNA的高斯相互作用谱核相似矩阵相结合。将已知的miRNA-疾病关联嵌入高维空间,训练两个模型以提取疾病和miRNA在高维空间中的表示。然后,提出了一个7层深度稀疏自编码模型,用于从已知的miRNA-疾病关系中挖掘潜在的miRNA-疾病关系。基于交叉验证的实验和比较其他案例研究的结果表明,DSAEMDA是有效和可靠的,并且优于几种最先进的方法。
几个关键因素促成了DSAEMDA的卓越性能。首先,DSAEMDA可以通过整合疾病相似性的学习算法提取疾病的高维特征表示,并将其嵌入到高维空间中。通过将疾病的两种语义相似度与疾病的高斯相互作用谱核相似度相结合,通过反向传播最小化损失函数来学习疾病的高维密集向量特征。miRNA的功能相似性结合miRNA的高斯相互作用谱核相似性和通过反向传播最小化损失函数学习到的miRNA的高维密集向量特征。其中,我们使用随机梯度下降(Adam)程序寻找最优解,以保证miRNA特征向量和疾病特征向量的可靠性。其次,我们为自编码器添加了稀疏性,在重构误差上添加了稀疏性惩罚,以限制隐藏层中每一层的所有神经元都被激活,这有利于特征提取。鉴于上述情况,与之前提出的方法相比,DSAEMDA确实提高了预测精度。
对于DSAEMDA,还有进一步改进的空间,例如网络层次结构和网络中的神经元数量。未来,随着更多样本的可用,我们将整合来自HMDDv3.2数据的更多miRNA-疾病关联来训练DSAEMDA。我们将结合各种神经网络,使用具有更深层次或更复杂模型结构的神经网络架构来实现更好的性能。

