1. 引言
肝脏是构成人体生命所需的重要器官,肝癌是世界上最常见的癌症之一。根据世界卫生组织发布的最新数据显示,每年都会有大量的病人因为肝癌疾病而死亡 [1]。在医学上,筛查肝脏疾病的主要手段是通过计算机断层扫描(Computed Tomography, CT)形成具有多个连续的层面图像,然后由放射科医生手工或半手工地来勾画出肝脏或病变区域,以作进一步的诊断及治疗计划 [2]。但是,手工或半手工地来勾画出肝脏或病变区域是非常耗时的,并且带有强烈的主观意志,容易出错。因此,肝脏与肿瘤的自动分割技术成为当前一个热点研究课题。
在CT图像中,肝脏与周围器官及组织之间的对比度非常低,从CT中自动、鲁棒地分割肝脏与肿瘤是一项非常具有挑战性的任务。而与肝脏分割相比,肿瘤具有形状和大小高度可变的特点,因此,肿瘤分割被认为是一项更具挑战性的任务。
近年来,深度学习技术已被广泛应用在医学图像的分割任务中,越来越多的研究者在肝脏与肿瘤分割任务中使用了深度学习方法 [3] [4] [5]。目前,最先进的肝脏与肿瘤分割方法大多是基于全卷积神经网络(Fully Convolutional Networks, FCN) [6]。FCN的核心主要是基于编码–解码的结构,对图像进行像素级分类,其可以输入任意大小的图像并产生相同大小的输出。基于FCN,Ronneberger等人 [7] 提出一个完全对称的U形网络,称为UNet,其主要创新是每一次上采样都与对应的特征提取部分进行融合,以弥补更多的低级语义信息,并且只需用少量的数据进行训练就能获得比较精确的结果。但是,该网络的缺点是,下采样操作会丢失大量的语义信息。为解决这个问题,Liu等人 [8] 在UNet的基础上,设计了适用于三维医学图像分割的网络,称为VNet,引入了ResNet的残差连接思想 [9],从编码器提取肝脏特征,解码器产生全分辨率输出。该方法虽然提高了分割精度,但由于三维卷积的参数量大,需要更长的训练时间。为此,Li等人 [10] 提出H-DenseUNet来缓解这个问题,其思路是混合2D DenseUNet与3D DenseUNet的特征来加速3D DenseUNet的收敛,同时在UNet中嵌入密集连接块,能进一步提升肝脏与肿瘤的分割精度。进一步地,Han等人 [11] 提出以多个2D信息堆叠成3D信息进行肝脏与肿瘤的分割,该方法基于UNet的跳跃连接和ResNet的残差连接,然后通过输入多个连续的单张切片来提供3D上下文信息。但是,这种方法得到的3D上下文信息是有限的。此外,为了分割多尺度的肿瘤特征,Jin等人 [12] 提出RA-UNet对肝脏和肿瘤进行分割,其以3D UNet为基准网络,在网络的深层引入了注意力机制来实现肝脏与肿瘤的分割,虽然能在一定程度上提升分割精度,但是由于深层的特征越来越抽象,语义信息非常少,因此提升是非常有限。相似地,Chen等人 [13] 提出Deeplabv3+模型。该模型是在空洞卷积和空间金字塔(ASPP)的基础上 [14],将深度可分卷积应用在ASPP和解码模块中,以此来学习更多的尺度特征。该方法在肿瘤的分割效果有进一步的提升,但在肝脏分割中,提升不明显。为此,Tang等人 [15] 提出两阶段的分割模型,首先用Res2Net50 [16] 作为基准粗分割出肝脏区域,再通过粗分割出来的肝脏区域裁剪输入,使用E2Net进行肝脏和肿瘤的精细分割。在精分割阶段,通过距离变换得到边缘增强监督信息,从而提高分割精度。
总的来说,以上方法大多都是基于UNet及ResNet的残差连接来实现肝脏与肿瘤的分割,证明了ResNet具有较强的特征提取能力。受此启示,本文直接采用ResNet作为特征提取网络。但是,上述方法中都忽视了三维医学图像的像素距离信息。如果CT图像的横断面的像素距离过大,直接采用3D卷积操作反而会带来很多干扰信息 [17],如图1所示,相邻横断面(xy)之间的像素距离约为6毫米。此外,上述方法难以拟合形状大小高度可变的肿瘤中的多尺度特征。针对第一个问题,本文提出改进的2.5D ResNet34在网络浅层学习单张切片的特征,在网络的深层学习整个图像的空间上下文信息,提高模型对三维空间信息的建模能力。针对第二个问题,本文提出尺度注意力感知模块,通过建模不同尺度特征下的局部和全局三维空间信息,以有效地整合低级上下文信息和高级区域语义信息,从而实现精准的肝脏和肿瘤分割。

Figure 1. Example of pixel distance among cross-sections of 3D medical images
图1. 3D医学图像横断面间像素距离示例图
2. 肝脏与肿瘤分割算法介绍
2.1. 整体结构
本文提出一种基于2.5D网络和尺度注意力感知模块的肝脏与肿瘤分割模型,该模型由三部分组成:1) 融合单张切片特征和相邻切片特征的2.5D ResNet34作为编码器;2) 尺度注意力感知模块;3) 多层解码单元组成的解码器,如图2所示。其中,与常用的ResNet34不同的是,本文在ResNet34的前两层改用2.5D卷积操作,在后三层采用正常的3D卷积操作,以缓解三维医学图像普遍存在的像素距离过大的问题。经过编码部分对输入图像进行编码后,产生四个不同尺度的特征向量
,
。然后,尺度注意力感知模块(SAA)用于捕获
的注意力特征,并通过将高层级与底层级的注意力特征融合,形成具有尺度感知的注意力特征
。SAA不仅可以缓解由于下采样操作导致肿瘤边界难以精准分割的问题,还能解决由于肿瘤形状高度可变而导致的误分割问题。解码器旨在融合不同层级的
,最终输出与输入图像大小相同的分割概率图。
2.2. 编码器
编码器使用ResNet34作为骨干网络,它具有较好的特征提取能力且参数量适中,非常适用于3D图像的语义分割。然而,直接将3D图像输入到3DResNet34中进行训练是不合理的。现有的神经网络无法学习到像素间的距离信息,如果像素距离过大,直接执行3D卷积操作反而难以学习三维的空间信息。
为缓解这一问题,提出改进后的2.5D ResNet34作为编码器,使编码器能在相邻切片像素距离过大的条件下,仍然能同时有效地学习单张切片特征和相邻切片特征。编码器的具体细节如图3所示,为有效提取单张切片的特征,将原3 × 3 × 3卷积核替换为1 × 3 × 3卷积核,称为2.5D方法。相应地,padding设为(0, 1, 1)。一般地,网络的浅层使用2.5D方法,网络的深层使用3D方法能更好地平衡模型对单张切片特征和相邻切片特征的学习能力。因此,提议的方法只在前两层使用2.5D方法,其他层使用3D方法。
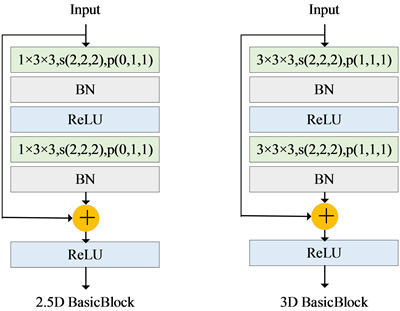
Figure 3. 2.5D basic block and 3D basic block structure diagram
图3. 2.5D基础模块和3D基础模块结构图
2.3. 尺度注意力感知模块
为了学习有效上下文信息,区域语义信息和边界信息在内的从粗到细的特征,本文提出了尺度注意力感知模块(SAA)。SAA的目标是探索不同尺度特征下的局部和全局三维空间信息。构造SAA的灵感来自RFB [18],其旨在通过卷积和扩展卷积的无缝组合来获得全局信息。与RFB把不同的是,SAA不仅引入了注意力机制,而且还根据不同的尺度建立了不同的分支,以进一步促进视觉感受野。具体地,SAA形成具有不同尺度的多分支注意力特征池。然后,嵌入多尺度特征融合策略,以有效地整合低级上下文信息和高级区域语义信息。
图4展示了尺度注意力感知模块的所有细节。假设编码器生成的多尺度特征为
,
,我们将不同尺度特征下的视觉感知过程表述如下:
(1)
(2)
(3)
其中,
表示获得的中间特征,
表示上采样操作,
表示softmax激活操作,
表示单尺度的注意力特征,
表示第s个尺度注意力特征。r表示空洞卷积率,
。

Figure 4. Structure diagram of scale attention awareness module
图4. 尺度注意力感知模块结构图
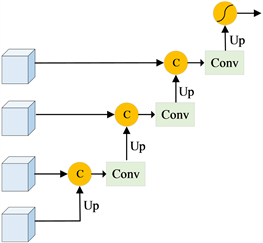
Figure 5. Diagram of the decoder module structure
图5. 解码模块结构图
2.4. 解码模块
解码模块主要由四层解码单元组成,如图5所示。每一层解码单元都是对高层级的尺度注意力感知特征进行上采样,得到与上一层级特征大小一致的新特征,然后将这两个大小一致的特征进行拼接融合,以弥补由于下采样操作中丢失的语义信息,从而增强对边缘细节的分割。最后,在最后一层解码单元中输出的特征向量被输入到softmax激活函数中,将特征向量映射成概率值,对应图像中每一个像素点,并选择概率最大的类别作为预测结果。
2.5. 损失函数
交叉熵损失函数是目前语义分割任务中使用最广泛的损失函数。但是,在肝脏与肿瘤分割任务中,将面临着类别不平衡问题,直接使用交叉熵损失函数会使模型倾向拟合类别数量多的样本。目前,加权的交叉熵能有效的缓解这类问题,其主要思想是对每个类别设置一个权重系数,对数量少的类别加大惩罚,公式如下所示:
(4)
其中,N表示图像的所有像素点的数量,c表示类别(背景,肝脏和肿瘤),
表示权重,
表示第i个像素点的真实值,
表示第i个像素点被预测为类别c的概率值。
对于语义分割任务,加权交叉熵只是在像素级计算损失值,忽视了整体结构的损失值。因此,引入了Dice相似性系数(Dice similarity coefficient, DSC)作为辅助损失函数。DSC用于度量两个集合的相似性,是评估分割精度最常用的度量指标,公式如下所示:
(5)
其中y表示真实值,p表示预测概率值。DSC损失函数可以定义为:
(6)
最后,我们使用联合损失函数用于肝脏与肿瘤分割,能够缓解类别不平衡的问题的同时,强调整体结构的损失值。公式如下所示:
(7)
其中,
为DSC损失函数的调整权重系数。
3. 实验与结果分析
3.1. 数据集及预处理
数据集是从国际医学图像计算和计算机辅助干预协会(Medical Image Computing and Computer Assisted Intervention Society, MICCAI)在2017年发起的肝脏肿瘤分割竞赛中获得的(Liver Tumor Segmentation, LiTS) [19]。LiTS包括来自6个不同医院的200个对比增强的3D腹部CT扫描图像,其中训练样本包含130个病例,测试样本包含70个病例。为了进行有监督的训练,MICCAI提供了由有经验的放射科医生对肝脏和肿瘤进行手工勾画的掩码图及注释。数据集在图像质量、空间分辨率和视觉方面存在显著差异,平面内分辨率固定为512 × 512像素,像素距离为0.6 mm × 0.6 mm~1.0 mm × 1.0 mm。切片间距离为0.45~6.0 mm,不同病例的切片数量为42~1026张不等。
与自然图像相比,CT扫描图像中存储的像数值单位为霍斯菲耳德单位(Hounsfield unit, Hu),像素值范围在[−3000, 3000]之间。在本实验中,通过分析CT图像和肝脏区域的窗宽和窗位值得到[−200, 250]内为最合适的强度值。所以,把大于250 Hu的像素设为250,小于−200 Hu的像素设为−200,从而更清晰观察到需要分割的肝脏区域 [20]。图6展示了原始CT图和经过窗宽和窗位调整后的对比效果,由图中可以观察到,经过窗宽和窗位调整后,能更清晰地观察到肝脏区域。
在数据集划分方面,本文把130个病例按照8:2的比例随机划分为训练集和验证集,剩下的70例作为测试集。此外,为防止模型过拟合,本文通过随机裁剪的方式对原图进行裁剪,裁剪后的大小为32 × 256 × 256,同时进行上下和左右翻转来达到数据扩充的目的。
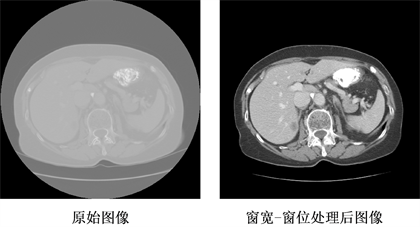
Figure 6. Comparison before and after of CT image processing
图6. CT图像处理前后对比图
3.2. 评价指标
为了评估模型在肝脏与肿瘤分割的有效性,本文采用以下三种评价指标。
1) Dice相似度系数(Dice similarity coefficient, DSC)表示预测结果p与真实结果y之间的重叠率,取值范围0~1,0表示预测结果与真实结果发生严重偏离,1表示预测结果与真实结果完全重合。其公式如公式5所示。
2) VOE (volumetric overlap error)表示错误率,单位为%,0%表示完全分割,100%表示预测结果与真实结果完全不重叠。其公式为:
(8)
3) ASD (average symmetric surface distance)表示预测结果与真实结果之间对称位置的平均表面距离,单位是mm,其中0 mm表示完美分割。其公式为:
(9)
其中,
代表集合x中的表面体素,
表示任意体素v到
的最短距离。
3.3. 实验参数设置
在参数设置上,初始学习率为0.001,同时利用权重衰减为1e−5的自适应矩估计优化器来实现梯度下降。此外,总训练轮次为200 epoch,每50个epoch将学习率降低10%,并在每个epoch之后在验证集上评估性能。本文所有的实验都是在NVIDIA GTX 3090 GPU上进行的。
3.4. 消融实验
为验证本文提出的不同组件的有效性,本实验采用相同的参数和数据集,通过逐一添加组件进行对比,肝脏与肿瘤分割在测试集上的实验结果分别如表1和表2所示。

Table 1. Comparison of the results of different components in the liver segmentation
表1. 不同组件在肝脏分割的结果对比

Table 2. Comparison of the results of different components in the tumor segmentation
表2. 不同组件在肿瘤分割的结果对比
由表1和表2数据可知,相比2D ResNet34,3D ResNet34由于学习到三维的空间信息,因此有助于肝脏和肿瘤的分割,肝脏的DSC提升1.9%,肿瘤的DSC提升63%。进一步地,通过将加权交叉熵损失函数(LWCE)和DSC损失函数(LDSC)结合形成混合损失函数,在肝脏和肿瘤分割的DSC上能得到进一步的提升,证明这种组合对于肝脏和肿瘤分割任务能发挥更好的效果。2.5D ResNet34方法在优化了三维空间信息后,肝脏分割的DSC能提高至95.7%,ASD进一步优化至1.2 mm。虽然3D ResNet34同样具有三维空间信息,但由于图像横截面的像素距离大,过多的使用三维空间信息反而会带来差的效果,2.5D ResNet34方法同样被证明在肿瘤分割中有效,相比3D ResNet34,DSC提升1.6%。最后,通过嵌入尺度注意力感知模块(SAA),在肝脏分割中,DSC被进一步提升至96.4%,在肿瘤分割中,DSC被进一步提升至72.3%,证明了本文提出的方法在肝脏和肿瘤分割中的有效性。
图7和图8分别展示了各种组件对肝脏和肿瘤的推理结果,其中,绿色区域表示正确分割肝脏/肿瘤,红色区域表示该区域是肝脏/肿瘤,但模型预测为背景,蓝色区域表示该区域为背景,但模型预测为肝脏/肿瘤。由图可知,2D ResNet34由于没有利用三维的空间信息,无论是肝脏分割或者肿瘤分割,都有较多的漏分割的区域。相反,由于医学图像在横断面的像素距离较大,直接使用3D卷积操作导致出现较多的误分割区域。通过使用2.5D ResNet34能在一定程度上缓解这个问题。最后结合尺度感知注意力模块,本文最终的方法在肝脏分割中的效果达到最好,其漏分割和误分割的区域非常少,这也是这个方法的DSC最高,VOE和ASD最低的直接证明。
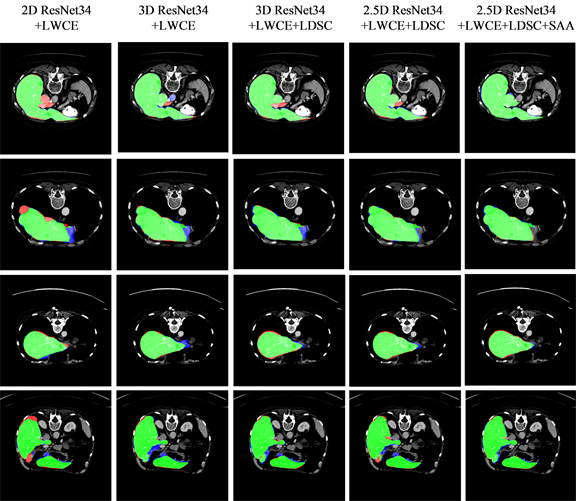
Figure 7. The inference results of liver among each of methods
图7. 各种方法对肝脏的推理结果
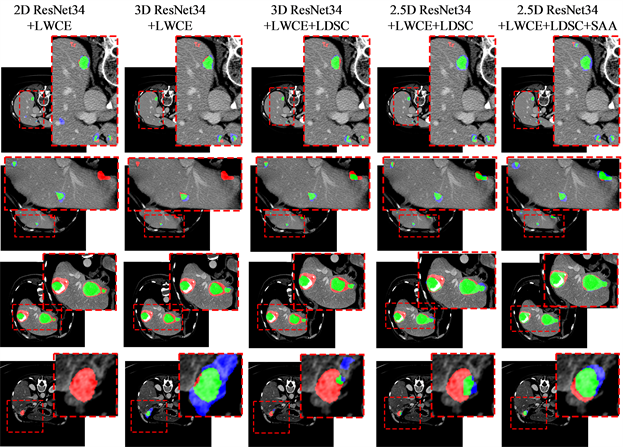
Figure 8. The inference results of tumor among each of methods
图8. 各种方法对肿瘤的推理结果
3.5. 对比实验
为进一步验证提出的方法的有效性,本文在LiTS的70例测试集上,比较了过去三年提出的肝脏与肿瘤分割方法以及一些主流的分割算法的DSC值。
UNet 2D:该模型是最先应用在医学图像的2D分割方法。其特点主要是在解码阶段,融合编码阶段的特征来弥补丢失的特性信息,被证明在医学图像分割中非常有效。
UNet 3D:由于UNet 2D不能充分学习三维的空间信息,因此衍生出3D版本。相比UNet 2D,UNet 3D能通过3D卷积操作来学习三维的空间信息,实现更准确的分割。
VNet:该模型主要是引入了残差连接弥补下采样的特征丢失语义信息的问题,并采用卷积替代上采样和平均池化,以保留更过细节信息。
DeeplabV3 + 3D:在空洞卷积和空间金字塔模块(ASPP)的基础上,将深度可分卷积应用在ASPP和解码模块中,提高网络的训练速度。
RA-UNet:该模型是一个三阶段分割模型,首先用RA-UNet 2D对肝脏进行定位,然后用RA-UNet 3D对定位的肝脏进行精细分割,最后用RA-UNet 3D从肝脏的感兴趣区域中分割肿瘤。
H-DenseUNet:该模型主要是在UNet的基础上加入了密集连接块来获取浅层的特征,同时通过融合2D DenseUNet和3D DenseUNet的特征进行精细的分割。
上述方法在LiTS的70例测试集的分割结果如表3所示。

Table 3. Comparison of the results of different components in the liver and tumor segmentation
表3. 不同方法在肝脏与肿瘤分割的结果对比
通过对比实验得出,本文提出的方法在肝脏和肿瘤分割中性能表现良好。其中,RA-UNet是通过使用两个模型来进行由粗到细的肝脏与肿瘤分割,而本文提出的是端到端的单模型分割方法。虽然多模型融合能有一定的DSC提升,但模型更复杂,且需要更多的推理时间。与RA-UNet相比,本文提出的方法在肝脏分割中,DSC提高了0.3%。在肿瘤分割中,DSC达到72.3%。同样地,H-DenseUNet的结果也是通过多个模型融合得到的,但本文的方法结果仍然比H-DenseUNet的更优,这显示了本文的方法在临床实践中具有更高效的应用潜能。
4. 结论
在肝脏与肿瘤分割任务中,针对三维医学图像的横断面间像素距离大的问题,本文基于2D和3D ResNet34,提出利用改进后的2.5D ResNet34对肝脏和肿瘤特征进行编码,提高模型对三维空间信息的建模能力。同时,利用DSC损失函数来提高模型对整体结构的分割能力。此外,针对肿瘤形状大小高度可变的问题,本文设计了尺度注意力感知模块来建模不同尺度特征下的局部和全局三维空间信息,以有效地整合低级上下文信息和高级区域语义信息。同时,在与其他先进的方法对比中,证明了本文提出的方法的有效性。在未来的工作中,将注重研究小目标的语义分割算法,以进一步提高肿瘤的分割性能。